BLASTのつかいかた(Web版とローカル版)
バイオロジーにおいて最もよく使用される配列検索ツール(BLAST)の解説をします。
例題として以下の遺伝子配列が何かというのを探索します。
例題
以下の配列が何かを同定せよ。ただし、ヒトゲノム由来の配列であることがわかっているとする。
> seq1
atgagcacagcaggaaaagtaatcaaatgcaaagcagctgtgctatgggagttaaagaaa
cccttttccattgaggaggtggaggttgcacctcctaaggcccatgaagttcgtattaag
atggtggctgtaggaatctgtggcacagatgaccacgtggttagtggtaccatggtgacc
ccacttcctgtgattttaggccatgaggcagccggcatcgtggagagtgttggagaaggg
gtgactacagtcaaaccaggtgataaagtcatcccactcgctattcctcagtgtggaaaa
tgcagaatttgtaaaaacccggagagcaactactgcttgaaaaacgatgtaagcaatcct
caggggaccctgcaggatggcaccagcaggttcacctgcaggaggaagcccatccaccac
ttccttggcatcagcaccttctcacagtacacagtggtggatgaaaatgcagtagccaaa
attgatgcagcctcgcctctagagaaagtctgtctcattggctgtggattttcaactggt
tatgggtctgcagtcaatgttgccaaggtcaccccaggctctacctgtgctgtgtttggc
ctgggaggggtcggcctatctgctattatgggctgtaaagcagctggggcagccagaatc
attgcggtggacatcaacaaggacaaatttgcaaaggccaaagagttgggtgccactgaa
tgcatcaaccctcaagactacaagaaacccatccaggaggtgctaaaggaaatgactgat
ggaggtgtggatttttcatttgaagtcatcggtcggcttgacaccatgatggcttccctg
ttatgttgtcatgaggcatgtggcacaagtgtcatcgtaggggtacctcctgattcccaa
aacctctcaatgaaccctatgctgctactgactggacgtacctggaagggagctattctt
ggtggctttaaaagtaaagaatgtgtcccaaaacttgtggctgattttatggctaagaag
ttttcattggatgcattaataacccatgttttaccttttgaaaaaataaatgaaggattt
gacctgcttcactctgggaaaagtatccgtaccattctgatgttttga
BLASTのつかいかた(Web版)
1. 公式ページへ飛ぶ
公式ページ:https://blast.ncbi.nlm.nih.gov/Blast.cgi
2. クエリとリファレンスの配列によって使うBLASTを選ぶ

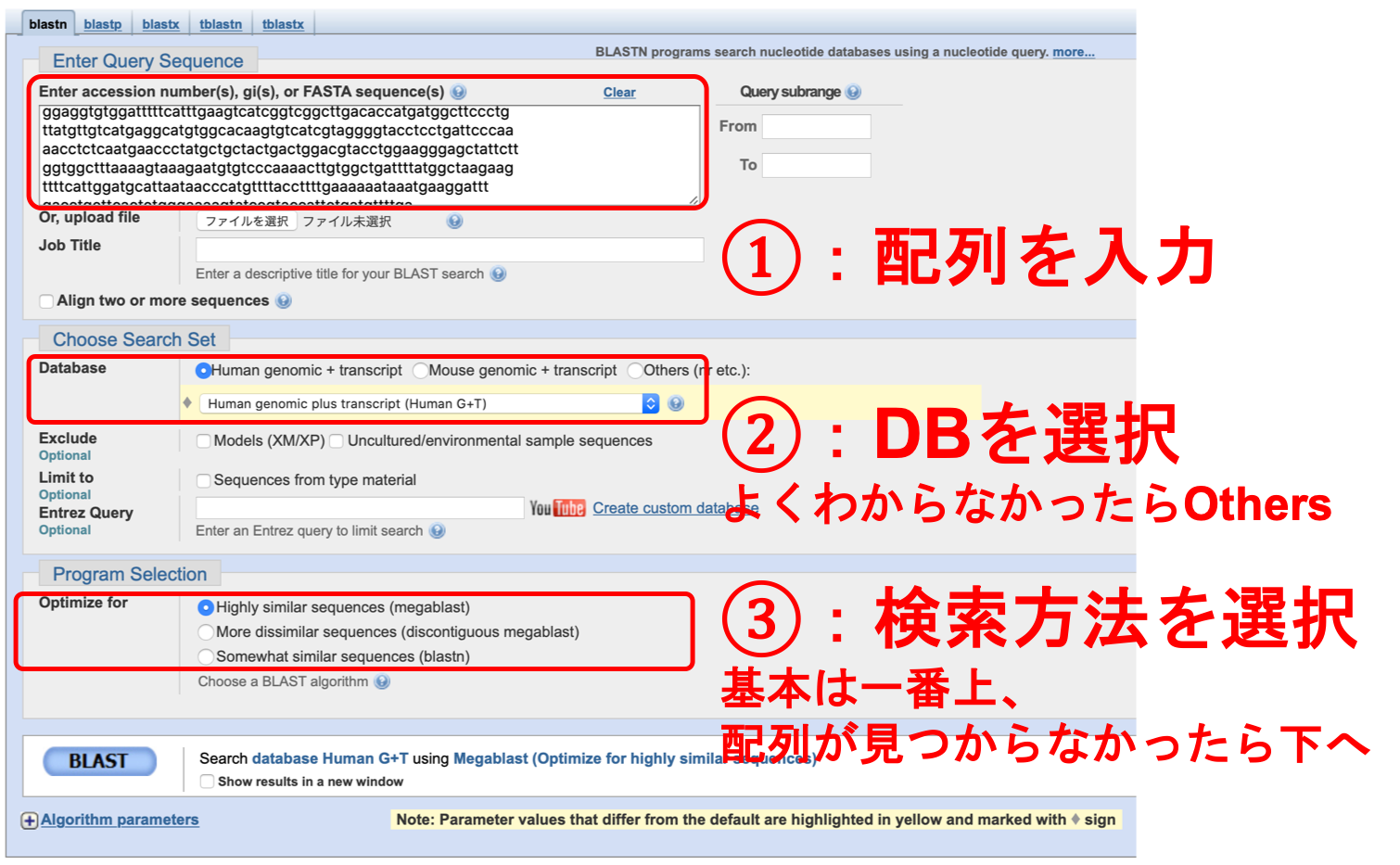
3. クエリ配列を入れた上で、オプション選んで検索
- Database
配列がヒト/マウス由来とわかっている場合はそれを、配列が何由来かわからない場合はOthersを選べばOK。
ただし、大きいDBほど機能未知の配列なども混じっているため注意すること。
| オプション | 説明 |
|---|---|
| Human genomic + transcript | ヒトゲノムなど |
| Mouse genomic + transcript | マウスゲノムなど |
| Others (nr etc.) | nrは全ての配列が入っているため、生物種不明のときはnrが無難。 |
- Program Selection
基本的にデフォルトのmegablastでOK。配列が見つからなかったら下のオプションを選ぶ。
| オプション | 説明 |
|---|---|
| Optimize for Highly similar sequences (megablast) | 類似配列があるとわかっている場合 |
| Optimize for More dissimilar sequences (discontiguous megablast) | マウスゲノムなど |
| Optimize for Somewhat similar sequences (blastn) | nrは全ての配列が入っているため、生物種不明のときはnrが無難。 |
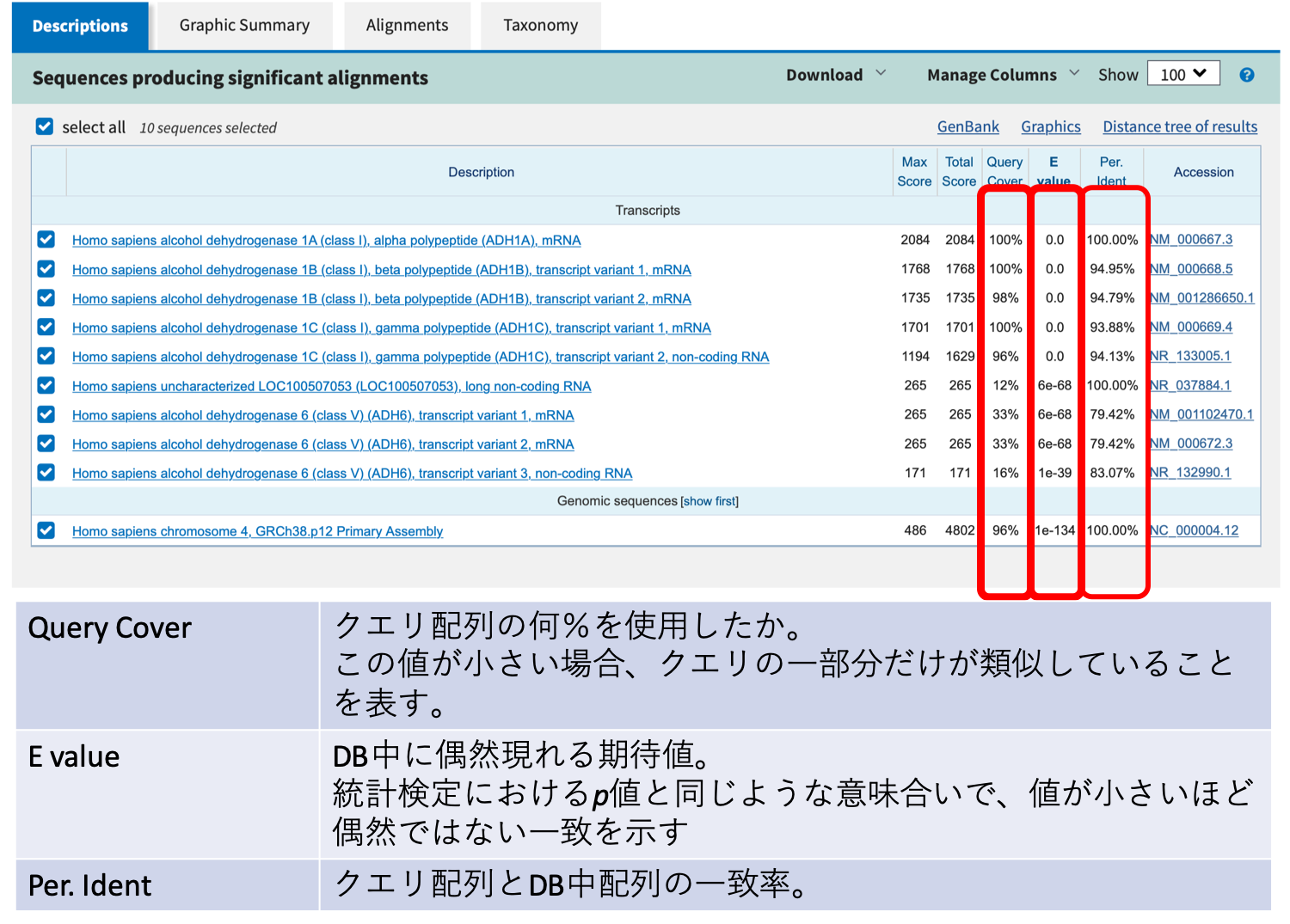
4. 結果を眺める
結果を眺めます。主に気をつけた方がいいのは、一致率(Per. Ident)とカバー率(Query Cover)です。
カバー率が低い場合、部分的な配列しか一致していません。
それでも問題ない場合もありますが、そこを考慮してください。
BLASTのつかいかた(ローカル版)
DBも自分で作った場合や、大量の配列検索をする場合はローカル版のBLAST(+)のインストールが必要になります。
0. ヒトゲノム配列の準備
ローカル版のBLASTの場合、Refrenceとなる配列も自分で準備することがあります。
今回はWeb版と同じく、ヒトゲノム遺伝子配列を準備します。
NCBIにあるヒトゲノムリファレンス配列のGRCh38(Genome Research Consortium human build 38)を使用します。
https://www.ncbi.nlm.nih.gov/genome/guide/human/
wget ftp://ftp.ncbi.nlm.nih.gov/refseq/H_sapiens/annotation/GRCh38_latest/refseq_identifiers/GRCh38_latest_rna.fna.gz
gunzip GRCh38_latest_rna.fna.gz
1. BLASTのインストール
公式ページのいうとおりにインストールしてみます。
BLASTのバージョンなどは変更する必要がありますが、以下のようなコマンドでインストールできます。
# $HOME/src にBLASTのtar.gzファイルなどを、
# $HOME/src/bin にコマンドをインストールします。
# mkdir -p $HOME/src
# mkdir -p $HOME/src/bin
# cd $HOME/src
# wget
wget ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.9.0+-src.tar.gz
# 解凍
tar -zxvf ncbi-blast-2.9.0+-src.tar.gz
# 移動
cd ncbi-blast-2.9.0+-src/c++
# ./configure && make && make install
./configure --prefix=$HOME/src
make
make install
2. DBのインデックス作成
BLASTで検索をかけるまえに、データベース配列の整理(インデックス作成)をする必要があります。
# アミノ酸配列の場合は -dbtype proc にする
makeblastdb -in GRCh38_latest_rna.fna -dbtype nucl -hash_index
GRCh38_latest_rna.fna のあとに3文字くらいついたファイル群ができていればOKです。
私の環境では以下のファイル群が作成されました。
$ ls -1 GRCh38_latest_rna.fna.*
GRCh38_latest_rna.fna.nhd
GRCh38_latest_rna.fna.nhi
GRCh38_latest_rna.fna.nhr
GRCh38_latest_rna.fna.nin
GRCh38_latest_rna.fna.nog
GRCh38_latest_rna.fna.nsd
GRCh38_latest_rna.fna.nsi
GRCh38_latest_rna.fna.nsq
3. 検索
上記の塩基配列をinput.faに入れ、blastしてみます。
# -outfmt 6 : タブ区切りで出力
blastn -db GRCh38_latest_rna.fna -outfmt 6 -query input.fa -out output.tsv
結果は以下のようになりました。
seq1 NM_000667.4 100.000 1128 0 0 1 1128 72 1199 0.0 2084
seq1 NM_000668.6 94.947 1128 57 0 1 1128 71 1198 0.0 1768
seq1 NM_001286650.2 94.789 1113 58 0 16 1128 208 1320 0.0 1735
seq1 NM_000669.5 93.883 1128 69 0 1 1128 72 1199 0.0 1701
seq1 NR_133005.2 94.133 784 46 0 1 784 72 855 0.0 1194
seq1 NR_133005.2 92.691 301 22 0 828 1128 855 1155 6.64e-120 435
seq1 NR_037884.1 100.000 143 0 0 119 261 3985 3843 9.22e-69 265
seq1 NM_001102470.2 79.420 379 74 2 376 752 470 846 9.22e-69 265
seq1 NM_000672.4 79.420 379 74 2 376 752 470 846 9.22e-69 265
seq1 NR_132990.2 83.069 189 31 1 564 752 300 487 2.07e-40 171
ベストヒット配列のIDで検索をかけると、Webの結果と同じくアルコールデヒドロゲナーゼであることがわかります。
$ grep "NM_000667.4" GRCh38_latest_rna.fna
>NM_000667.4 Homo sapiens alcohol dehydrogenase 1A (class I), alpha polypeptide (ADH1A), mRNA
参考
- 旧BLASTと新BLASTのオプション対応表
http://togotv.dbcls.jp/20110225.html
- E-valueの計算方法(DDBJ)
https://www.ddbj.nig.ac.jp/faq/ja/how-is-e-value-calculated.html
- E-valueの計算方法(NCBI・英語)
https://www.ncbi.nlm.nih.gov/BLAST/tutorial/Altschul-1.html
https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Web&PAGE_TYPE=BlastDocs&DOC_TYPE=FAQ
- E-valueの計算方法(わかりやすい) https://note.mu/tatanzaw/n/n82ed08c96c77