t-SNE
t-SNE(t-Distributed Stochastic Neighbor Embedding; t分布型確率的近傍埋め込み法)とは?
多変量解析の手法の1つです。
複数次元あるデータを要約して、データの関係性をわかりやすく要約してくれます。
よくある勘違いですが、クラスタリングと違い、データがどういうクラスターに属するかはわかりません。
クラスタ分けはk-meansなどで別途行う必要があります。
アルゴリズム・使用上の注意
アルゴリズムに関しては、Qiitaのページを参照ください。
一言でいうと、高次元の距離情報を2次元の距離情報に落としたときの損失を最小限にするような点の探索と解釈しています。
t-SNEの中で重要なパラメーターとして、perplexityというものがあります。
これは、各点の有効な近傍点の個数を表します。値が小さいほど、より似てるものとの関係性しか考慮しないということ。
このページでは、
-
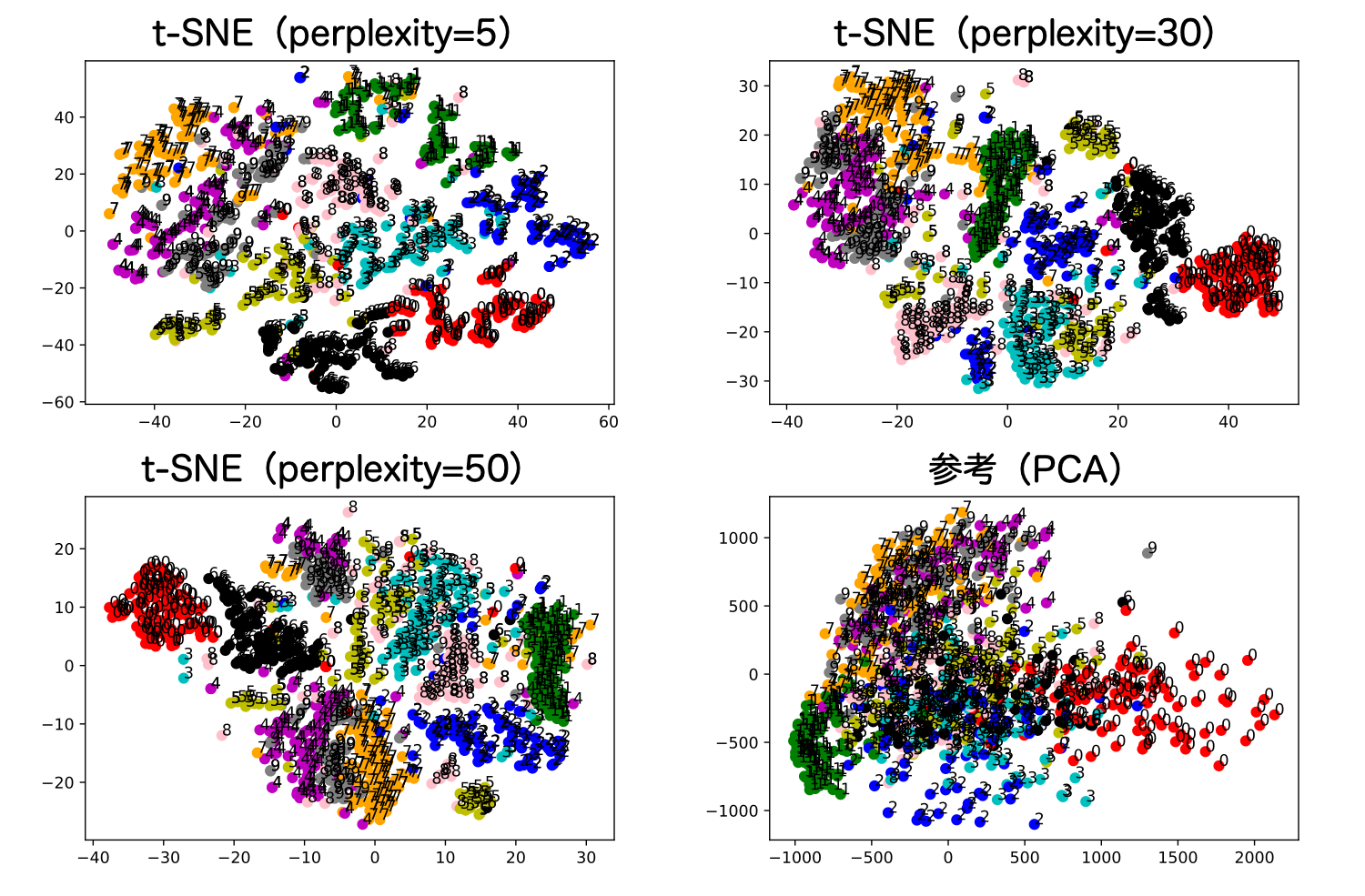
論文中で5〜50がいいと記載があること
-
RとPython両方のライブラリにおいてデフォルトの値が30であること
から、5, 30, 50の3つを探索するようにしています。
例題
画像データとして有名な、MNISTデータを用います。
CSVへ変換した結果を公開しているリポジトリがあるので、ここからデータをダウンロードします。
データ全体の可視化はこのあと取り扱っていきますので、どのような規則でデータが入ってるかを可視化してみます。
1行目にどのようなデータが入っているかを可視化します。
#!/usr/bin/env python
#!pip install japanize_matplotlib # プロット中で日本語を使用するためにインストール
import itertools
import pandas as pd
import matplotlib.pylab as plt
from IPython.display import display
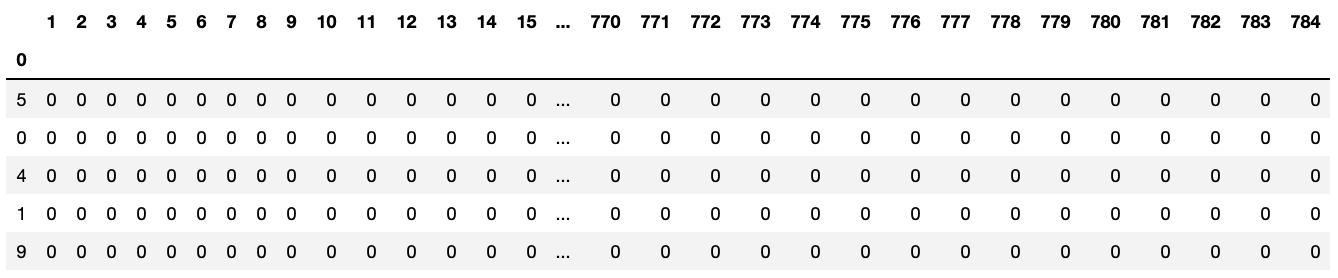
df = pd.read_csv("mnist_train.csv", header=None, index_col=0)
display(df.head())
xs = []
ys = []
for x, y in itertools.permutations(range(28), 2):
iloc = y * 28 + x
if df.iloc[0, iloc] != 0:
xs.append(x)
ys.append(28 - y)
plt.scatter(xs, ys)
# 出力
df.head()の出力結果
plt.scatter(xs, ys)の出力結果

本来、色の濃さを表す数値が各ビットに入っているのですが、一旦それを無視して可視化しても数字の「5」が書いてあることがなんとなくわかるかと思います。
ソースコード
# Pythonでt-SNE
Pythonの場合は scikit-learnでt-SNEを行うことができます。
以下では、trainデータのうち、1000行だけ計算しています。
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.manifold import TSNE
df = pd.read_csv("mnist_train.csv", header=None, index_col=0)
df_1000 = df.sample(1000)
# perplexityは30がデフォルトが多い(Rtsneもsklearnも30)
# 5〜50がよいと言われているため、5,30,50
colors = ["r", "g", "b", "c", "m", "y", "k", "orange", "pink", "gray"]
col = ["x", "y"]
for perplexity in [5, 30, 50]:
model = TSNE(n_components=2, perplexity=perplexity, n_iter=500, verbose=3, random_state=1)
transformed = model.fit_transform(df_1000)
df_transformed = pd.DataFrame(transformed, index=df_1000.index, columns=col)
df_transformed["color"] = [colors[i] for i in df_transformed.index]
plt.scatter(df_transformed["x"], df_transformed["y"], c=df_transformed["color"])
plt.savefig(f"scatter.tsne.{perplexity}.pdf")
plt.clf()
結果は以下のように出力されます。
# Rでt-SNE
Rでt-SNEの場合、Rtsneというライブラリを使用します。
ライブラリのタイトルにも記載がありますが、Barnes-Hut法により、高速化されたt-SNEを用いているようです。
#!/usr/bin/env Rscript
# install.packages("Rtsne") # インストール
library("Rtsne")
set.seed(0)
setwd("/Users/nsmt/prv/hp/py_r_stats")
# input
df = read.csv("mnist_train.csv", header=FALSE)
df_1000 = df[1:1000, ]
vec_ans = df_1000$V1
df_1000 = df_1000[, colnames(df_1000) != "V1"]
# main
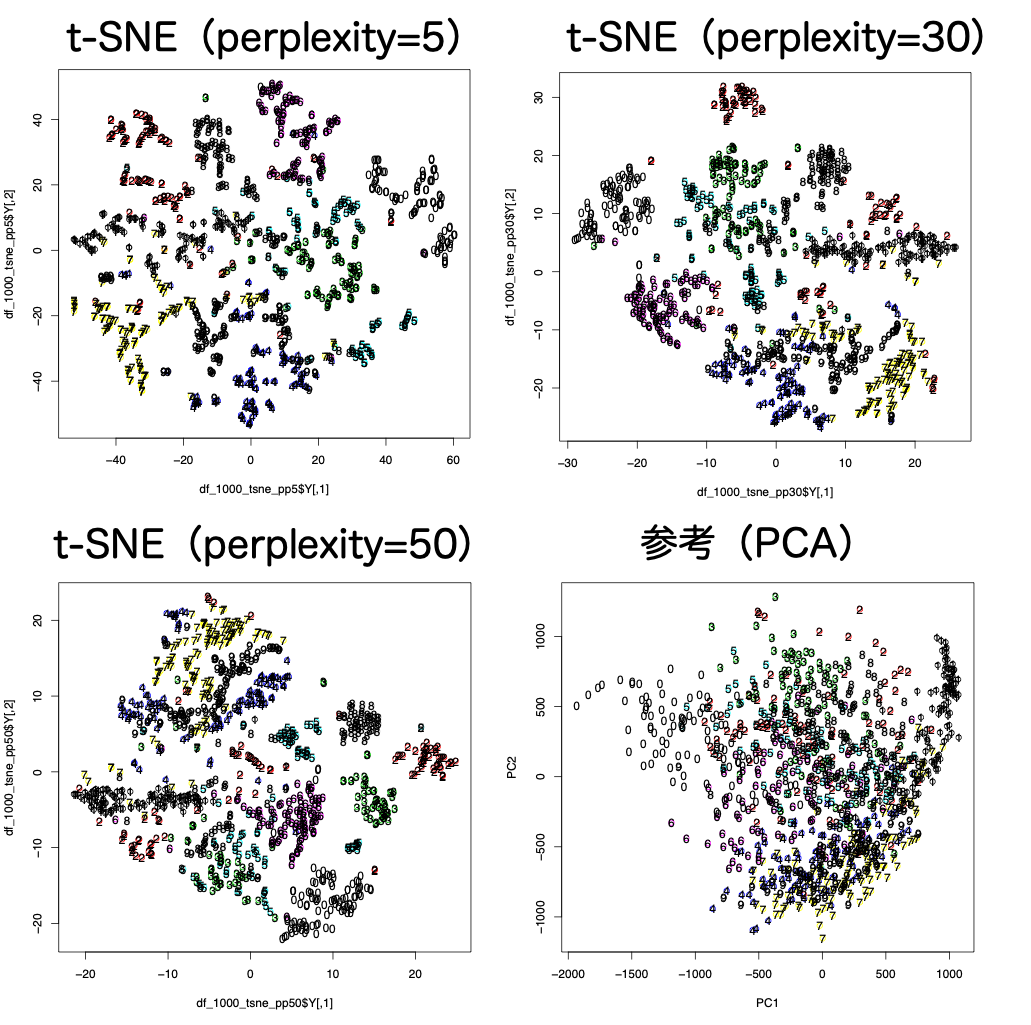
df_1000_tsne_pp5 = Rtsne(df_1000, perplexity=5)
df_1000_tsne_pp30 = Rtsne(df_1000, perplexity=30)
df_1000_tsne_pp50 = Rtsne(df_1000, perplexity=50)
# output
pdf("scatter.Rtsne.5.pdf")
plot(df_1000_tsne_pp5$Y, col=vec_ans)
dev.off()
pdf("scatter.Rtsne.30.pdf")
plot(df_1000_tsne_pp30$Y, col=vec_ans)
dev.off()
pdf("scatter.Rtsne.50.pdf")
plot(df_1000_tsne_pp50$Y, col=vec_ans)
dev.off()
結果は以下のように出力されます。
その他
比較に使用したPCAのソースコードを載せておきます。
- PCAのPythonコード
#!/usr/bin/env python
colors = ["r", "g", "b", "c", "m", "y", "k", "orange", "pink", "gray"]
pca = PCA(n_components=2, random_state=0)
df_pca = pd.DataFrame(pca.fit_transform(df_1000))
df_pca.columns = ["PC1", "PC2"]
df_pca["color"] = [colors[i] for i in df_1000.index]
- PCAのRコード
#!/usr/bin/env Rscript
df_1000_pca = prcomp(df_1000)
pdf("scatter.RPCA.pdf")
plot(df_1000_pca$x, col=vec_ans)
text(df_1000_pca$x[, 1], df_1000_pca$x[, 2], vec_ans)
dev.off()